DNA測序是一種生物學技術,用於確定DNA分子中鹼基的順序或序列。 DNA是生物體內攜帶遺傳信息的分子,其由一系列不同的鹼基(腺嘌呤、胞嘧啶、鳥嘌呤和胸腺嘧啶,也就是ATCG)組成。 DNA測序的主要目的是揭示DNA分子的基本組成,以便研究和理解生物學、遺傳學、進化和醫學等各個領域的問題。 DNA測序的基本原理是確定DNA鏈中鹼基的排列順序。
所以說DNA測序的基本原理是確定DNA鏈中鹼基的排列順序,DNA測序的歷史可以分為第一代、第二代和第三代,每一代都有其獨特的技術和特點。
第一代DNA測序的主要突破發生在1977年,當時發展出了Maxam-Gilbert和Sanger測序兩種方法。Sanger測序以其更高的準確性、穩健性和易用性而脫穎而出,迅速成為最常用的DNA測序技術,並在許多年內保持如此。這種方法也稱為鏈終止測序,它利用二氧化鏈終止核苷酸在DNA複製過程中的插入,從而確定序列。第一代測序的主要特點是其相對較低的通量,並且需要較長的時間來完成。然而,它在當時的DNA研究中起到了關鍵作用。
第二代測序始於20世紀90年代末,當時瑞典斯德哥爾摩皇家理工學院的一個研究小組引入了焦磷酸測序(pyrosequencing)。焦磷酸測序基於DNA合成過程中焦磷酸的釋放來進行測序。該技術於2005年由454 Life Sciences首次商業化,成為第一個商業化的下一代測序技術。然而,焦磷酸測序已被更先進的下一代測序方法所取代,例如Illumina測序。Illumina測序使用可逆終止的脱氧核苷酸(dNTPs)來確定序列,並自2007年首台測序儀推出以來經過了多次改進。Illumina測序現在是最廣泛使用的下一代測序技術之一,具有高通量和高效率的特點。
第三代測序是近年來的新興領域,旨在克服第二代測序技術的一些限制。第三代測序包括來自Pacific Biosciences(PacBio)和Oxford Nanopore Technologies(ONT)的兩種主要技術。PacBio測序監控了聚合酶酶在實時中的核苷酸插入,而Nanopore測序使用嵌入在脂質膜中的納米孔。當單鏈DNA分子通過這些孔時,可以檢測到根據分子的碱基序列而變化的電信號。第三代測序技術具有高通量、實時性和不需要DNA放大等優勢,但仍在不斷改進中。
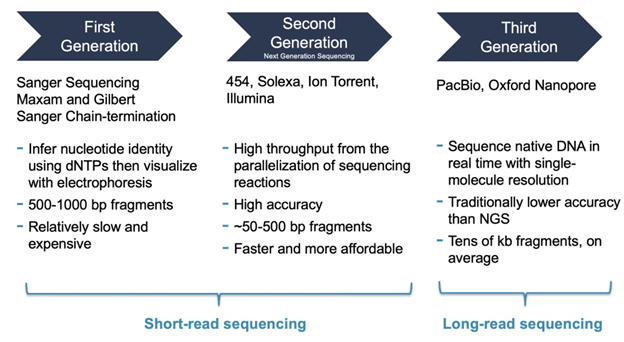
我們現在知道了歷史,來看這張圖就知道每代的差異了,最上面的一排,例如PacBio的那整行,都是一二三代的平台機器的名字。
其次下面的short read 與 long read 是指說測序的長短,因為人體的genome 基因體很長,所以我們會用「read」 也就是打碎的DNA片段,但因為技術的有限,所以二三代read長短不一樣。
首先一代的效率跟準確度真的不夠,所以我們就不再多提,二三代的差別其實也都在圖上指出,但為什麼現在大多數Biobank(跟生物相關的DB),所含有的datatype 都是NGS呢?
DNA測序歷史經歷了幾個關鍵的發展階段,從第一代的Sanger測序到第二代的高通量測序技術,再到近年的第三代測序技術。每一代都帶來了更高的效率、更大的通量和更低的成本,從而推動了DNA研究的不斷發展。
其實這邊我沒有講到怎麼知道DNA密碼的ATCG序列的,因為這一二三代的技術博大精深,有興趣跟想知道詳細差別的,可以看source 跟 youtube 找影片看差異。
DNA sequencing methods: from Sanger to NGS
